2025年9月17日,中国科学院大连化学物理研究所尹恒团队在《Proceedings of the National Academy of Sciences,PNAS》上在线发表研究论文“Characterization of SrUGT76G4 reveals a key residue for regioselectivity and efficient Reb M synthesis”。该工作报道了甜叶菊中负责莱鲍迪苷(Rebaudioside)D(Reb D)向莱鲍迪苷M(Reb M)转化的关键糖基转移酶UGT76G4,阐明了该酶对底物C19位区域选择性的分子机制,以及200位甘氨酸在此过程中的关键作用,并在此基础上进行了半理性化改造,获得了多个可用于高效合成Reb M的工具酶。
甜菊糖苷(Steviol glycosides) 是一类从甜叶菊中提取的天然甜味剂,其特点是甜度高(蔗糖的200-300倍),但热量极低,因此被广泛用作蔗糖的替代品。甜菊苷(Stevioside,STV)和瑞鲍迪苷(Rebaudioside)A在甜叶菊中含量最高,主要通过甜叶菊叶提取制备,生产工艺相对成熟,但有些微后苦味;与其他单体相比,瑞鲍迪苷D(Reb D)和瑞鲍迪苷M(Reb M)的甜度可达蔗糖的350倍,特别是Reb M,其甜味快速、干净,与蔗糖的口感更接近,且几乎没有后苦味,被称之为下一代甜叶菊甜味剂。但由于Reb M在甜叶菊中的含量极低,仅占叶片干重的0.4-0.5% w/w,加上受到作物生长周期的限制,因此从甜叶菊中直接提取的效率较低且成本高昂,产量难以满足市场需求。
目前,甜叶菊的改良育种和生物合成技术的应用是解决Reb M生产效率的主要方案。近年来,甜叶菊中甜菊糖苷的主要合成途径逐步得到了解析,而Reb D到Reb M转化的关键酶目前还没有完全明晰,比较普遍的看法是认为由UGT76G1催化完成。UGT76G1具有广泛的底物特异性,能作用于甜菊糖苷的C13-和C19-两端,催化多种甜菊糖苷的糖基化,其中包括催化Reb E到Reb D的转化以及Reb D到Reb M的转化。但是,近年来详细的酶动力学实验表明,UGT76G1具有显著的底物C13位区域选择性,其对底物C19位的催化活性很低,并且在过表达UGT76G1的甜叶菊植株中Reb M的含量并没有出现明显增加。
近年来,国内外科学家针对UGT76G1的催化机制及改造研究取得了一定的成果,但是所获得的突变体对C19位糖基化的催化效率仍普遍较低,远低于野生型酶对C13位的催化效率。此外,研究者们利用不同品种的甜叶菊和不同时期的植株中UGT76G1基因的多态性及随机等位基因表达性质进行了研究,发现UGT76G1天然突变体的活性及表达情况与植株中Reb A和Reb C的含量存在相关性。但是由于这些研究中所使用的甜叶菊品种中Reb M的含量都非常低甚至不含,从而限制了研究者们得到具有C19位高催化活性的UGT76G1天然突变体的可能性。
在实验室的前期研究中,尹恒课题组对浩天药业5个甜叶菊品种(其中包括3个高产Reb M的品种)进行了比较转录组分析,发现UGT76G家族基因确实与甜叶菊中Reb M合成密切相关(Plant Physiology and Biochemistry,2021, 167:541–549)。
在本研究中,作者根据前期获得的高产Reb M的甜叶菊转录组数据设计简并引物,从高产Reb M品系的植株中鉴定到13条突变体序列(UGT76G2至UGT76G14),并对UGT76G家族所有序列构建系统发育树(原文图1A)。将所有克隆到的UGT76G1天然突变体基因在大肠杆菌中进行了异源表达、纯化以及活性测定,发现UGT76G4具有显著的底物C19位催化活性以及区域选择性,并且这种偏好性在瞬时转化UGT76G4的烟草叶片中能同样观察到(原文图1B)。UGT76G4催化Reb E生成C19+C13双糖基化产物Reb M,中间产物为C19位糖基化的Reb AM(原文图1E,F),催化Rub生成双糖基化Reb E6,中间产物为C19位糖基化的STV B(文章附图),显著不同于UGT76G1。该结果表明甜叶菊中甜菊糖苷糖基化途径的多样性(原文图1G)。
进一步酶动力学实验表明UGT76G4对Reb E的C19位糖基化效率(kcat/Km)是对其C13位糖基化效率的3.6倍,而以Rub为底物是也的得到类似的结果,这进一步证明了UGT76G4对甜菊糖苷底物的C19位区域选择性。针对底物Reb D,UGT76G4的催化效率是UGT76G1的11.3倍,表明该酶在甜叶菊中Reb D向Reb M转化途径中的关键作用。
为了明确导致UGT76G4和UGT76G1不同底物区域选择性(C13/C19)的关键氨基酸残基,作者首先对两者之间的差异残基进行了分析,发现差异残基主要集中在191-208区段,分布于底物结合口袋周围(原文图2A,B)。于是作者将191-208区段进行了互换,发现互换实现了两个酶之间区域选择性几乎完全的逆转(原文图2C,D)。为了进一步确认关键残基的位置,作者将UGT76G4中该区段内的差异残基逐个替换成UGT76G1中的残基,发现只有突变体G200L的底物区域选择性出现逆转,变成C13位偏好性(原文图2E)。为了明确200位不同的氨基酸组成对UGT76G4底物区域选择性的影响,作者对该位点进行了饱和突变,发现除组氨酸外,其他氨基酸都会导致区域选择性向C13转变(原文图2F)。这些结果证明了200位残基在调控UGT76G4区域选择性中的关键作用。有趣的是,将UGT76G1中的L200突变成甘氨酸并不能实现区域选择性向C19的逆转,表明200位的调控作用需建立在191-208其他氨基酸残基组成的基础上。
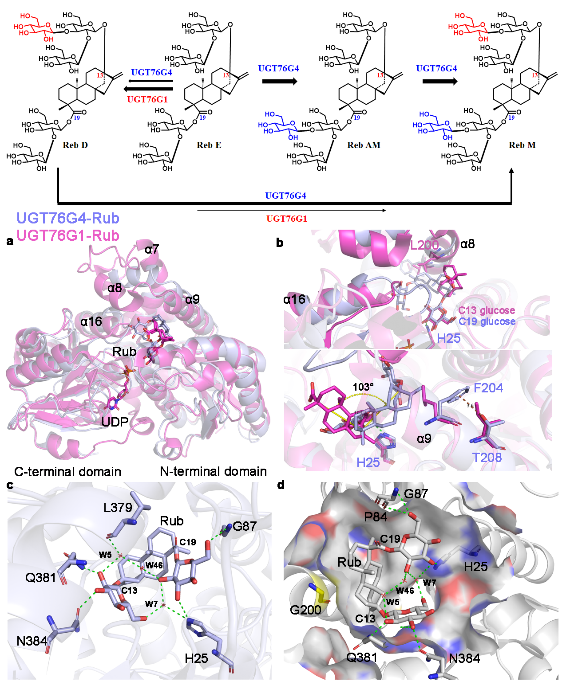
为了阐明UGT76G4不同于UGT76G1的底物区域选择性的分子机制,作者解析了UGT76G4-UDPG和UGT76G4-Rub(悬钩子苷,Rubusoside)的晶体结构(原文图3),发现与之前文献报道的UGT76G1-Rub晶体结构中Rub所呈现“竖直”状态的构象不同,Rub在UGT76G4的底物结合口袋内折叠成U型,呈“平躺”状态,与UGT76G1复合体中的Rub成103度旋转,且C19位糖基处于催化位置,与催化残基His25形成氢键。Rub在UGT76G4底物结合口袋采取这种构象主要是因为G200残基没有侧链,增加了空腔内部空间大小。
在UGT76G4晶体结构信息的基础上,作者通过分子动力学模拟进一步分析了UGT76G4及其突变体G200L结合不同构象Rub(C19催化位和C13催化位)时的动态相互作用(原文图4),发现由于200位残基(甘氨酸/亮氨酸)侧链的大小,结合Rub的C13位醚键和C19位酯键可旋转角度的差异,导致只有在UGT76G4-UDPG-Rub19和G200L-UDPG-Rub13这两种组合的复合体中,Rub才能处于正常的催化状态。从而解释了UGT76G4底物区域选择性的分子机制。
针对200位对别的植物UGT底物选择性是否具有普遍性,作者也进行了探索,发现将水稻来源的UGT91C1中对应位置(L204)突变成甘氨酸后,确实实现了其底物区域选择性的逆转(原文图5)。
为了进一步提升UGT76G4的C19位选择性及催化活性,作者基于前面获得的信息进行了半理想改造,最后获得了组合突变体Q199I/G200Y和H155S/Q199I/G200Y,对Reb E和Reb D的转化效率较UGT76G4分别提高了1.46倍和23倍(原文图6)。
总结而言,作者鉴定并表征了UGT76G1的一个天然突变体UGT76G4,UGT76G4在体内和体外实验中均表现出对甜菊糖苷底物独特的C19位区域选择性,是甜叶菊中Reb D到Reb M生物合成的关键酶。作者通过定点/饱和突变、X射线晶体学、分子动力学(MD)模拟等方法,阐明了UGT76G4的底物区域选择性的分子机制,明确了G200在决定UGT76G4的C19区域选择性中的关键作用。基于这些发现,作者对UGT76G4进行了工程改造,得到了Q199I/G200Y和H155S/Q199I/G200Y突变体,它们目前从Reb E和Reb D合成Reb M具有优势。
我组已毕业博士王宇和李唐副研究员为本文的共同第一作者,尹恒研究员为论文的通讯作者,山东省天然产物代谢工程与合成生物学工程研究中心相关人员参与了本项目研究。该工作及相关研究得到了浩天药业的持续支持,项目相关研究成果已获专利授权并转化该企业。
论文链接:https://www.pnas.org/doi/10.1073/pnas.2504698122
所新闻链接:https://www.dicp.ac.cn/xwdt/kyjz/202509/t20250923_7974871.html